MIT与哈佛联合发布Lyra:生物序列建模领域的重大突破
在计算生物学领域的一项重大进展中,来自MIT、哈佛大学和卡内基梅隆大学的研究人员推出了Lyra,这是一种突破性的生物序列建模方法。这一创新解决了生物学深度学习应用中长期存在的高计算成本和大数据集依赖的挑战。
效率的飞跃
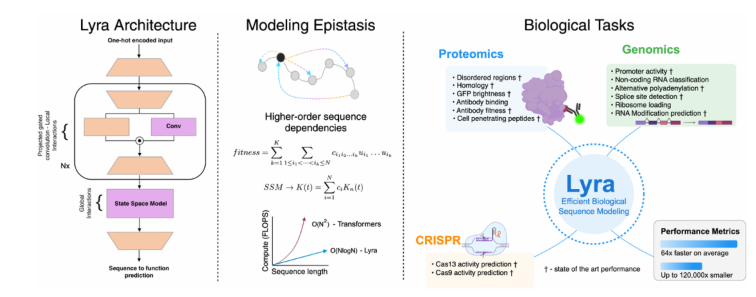
Lyra代表了生物序列分析领域的范式转变,仅需传统模型参数的12万分之一。值得注意的是,该系统可以仅用两块GPU在两小时内完成完整训练,使得资源有限的实验室也能进行高级生物研究。
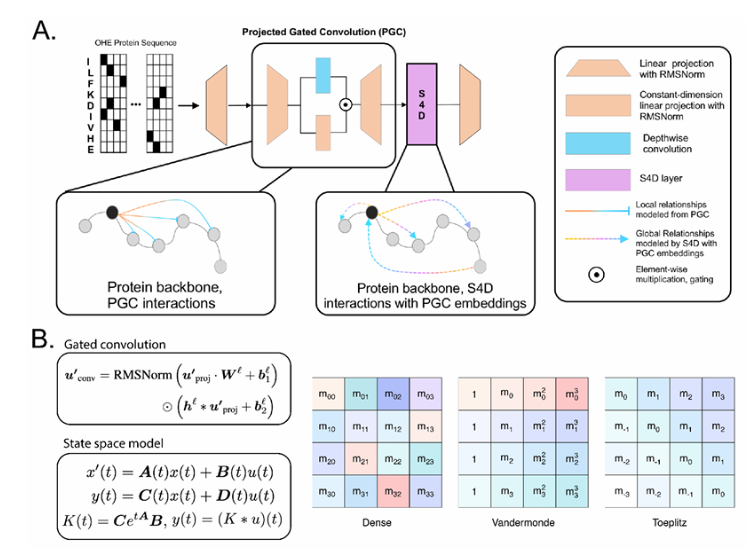
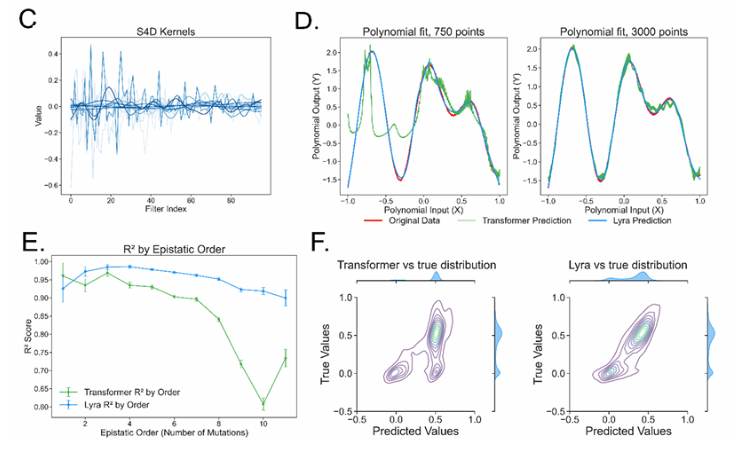
该模型的设计灵感来自上位性(epistasis)——序列内突变之间的相互作用。通过采用次二次架构(sub-quadratic architecture),Lyra有效解读了生物序列与其功能之间的复杂关系。
在多个应用中的卓越表现
在超过100项生物任务的测试中,Lyra在以下方面表现出色:
- 蛋白质适应性预测
- RNA功能分析
- CRISPR设计
该系统在多个关键应用中实现了最先进的性能(SOTA),在使用显著更少计算资源的同时超越了传统方法。
技术创新
研究团队开发了一种新颖的混合架构(hybrid architecture),结合了:
- 状态空间模型(SSM)用于通过快速傅里叶变换(FFT)进行全局关系建模
- 投影门控卷积(PGC)用于局部特征提取
这种创新方法使得推理速度比传统的卷积神经网络(CNN)和Transformer模型快64.18倍。
对生物研究的广泛影响
Lyra的高效性对多个领域产生了深远影响:
- 加速治疗开发
- 增强病原体监测能力
- 改进生物制造流程
研究团队希望他们的突破能够普及高级生物序列建模技术,使更多科学家能够在无需庞大计算基础设施的情况下为前沿发现做出贡献。
关键要点
- Lyra将模型参数减少至传统方法的12万分之一
- 仅用两块GPU在两小时内完成完整训练
- 在100多项生物任务中展示最先进性能
- 采用混合SSM-PGC架构以实现最佳效率
- 推理速度比传统CNN和Transformer快64.18倍



